如何实现并应用决策树算法?
本文对决策树算法进行简单的总结和梳理,并对著名的决策树算法ID3(Iterative Dichotomiser 迭代二分器)进行实现,实现采用Python语言,一句老梗,“人生苦短,我用Python”,Python确实能够省很多语言方面的事,从而可以让我们专注于问题和解决问题的逻辑。
根据不同的数据,我实现了三个版本的ID3算法,复杂度逐步提升:
1.纯标称值无缺失数据集
2.连续值和标称值混合且无缺失数据集
3.连续值和标称值混合,有缺失数据集
第一个算法参考了《机器学习实战》的大部分代码,第二、三个算法基于前面的实现进行模块的增加。
决策树简介
决策树算法不用说大家应该都知道,是机器学习的一个著名算法,由澳大利亚著名计算机科学家Rose Quinlan发表。
决策树是一种监督学习的分类算法,目的是学习出一颗决策树,该树中间节点是数据特征,叶子节点是类别,实际分类时根据树的结构,一步一步根据当前数据特征取值选择进入哪一颗子树,直到走到叶子节点,叶子节点的类别就是此决策树对此数据的学习结果。下图就是一颗简单的决策树:
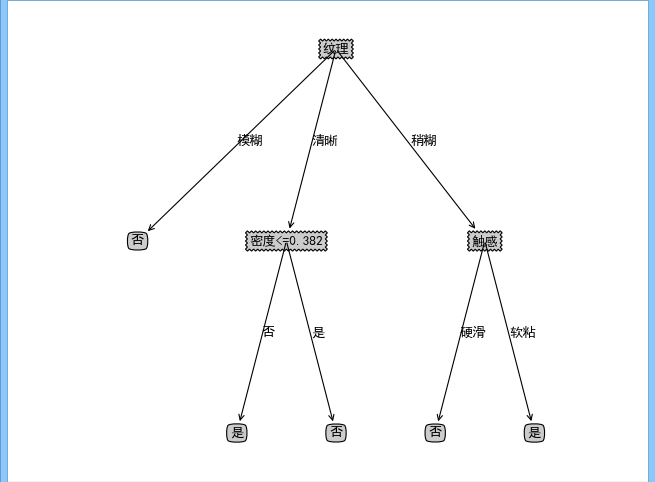
此决策树用来判断一个具有纹理,触感,密度的西瓜是否是“好瓜”。
当有这样一个西瓜,纹理清晰,密度为0.333,触感硬滑,那么要你判断是否是一个“好瓜”,这时如果通过决策树来判断,显然可以一直顺着纹理->清晰->密度<=0.382->否,即此瓜不是“好瓜”,一次决策就这样完成了。正因为决策树决策很方便,并且准确率也较高,所以常常被用来做分类器,也是“机器学习十大算法”之一C4.5的基本思想。
学习出一颗决策树首要考虑一个问题,即 根据数据集构建当前树应该选择哪种属性作为树根,即划分标准?
考虑最好的情况,一开始选择某个特征,就把数据集划分成功,即在该特征上取某个值的全是一类。
考虑最坏的情况,不断选择特征,划分后的数据集总是杂乱无章,就二分类任务来说,总是有正类有负类,一直到特征全部用完了,划分的数据集合还是有正有负,这时只能用投票法,正类多就选正类作为叶子,否则选负类。
所以得出了一般结论: 随着划分的进行,我们希望选择一个特征,使得子节点包含的样本尽可能属于同一类别,即“纯度”越高越好。
基于“纯度”的标准不同,有三种算法:
1.ID3算法(Iterative Dichotomiser 迭代二分器),也是本文要实现的算法,基于信息增益即信息熵来度量纯度
2.C4.5算法(Classifier 4.5),ID3 的后继算法,也是昆兰提出
3.CART算法(Classification And Regression Tree),基于基尼指数度量纯度。
ID3算法简介
信息熵是信息论中的一个重要概念,也叫“香农熵”,香农先生的事迹相比很多人都听过,一个人开创了一门理论,牛的不行。香农理论中一个很重要的特征就是”熵“,即”信息内容的不确定性“,香农在进行信息的定量计算的时候,明确地把信息量定义为随机不定性程度的减少。这就表明了他对信息的理解:信息是用来减少随机不定性的东西。或者表达为香农逆定义:信息是确定性的增加。这也印证了决策树以熵作为划分选择的度量标准的正确性,即我们想更快速地从数据中获得更多信息,我们就应该快速降低不确定性,即减少”熵“。
信息熵定义为:
$$Ent(D) = -/sum_{k=1}^{|/mathcal{Y}|}p_k/log_2p_k$$
D表示数据集,类别总数为|Y|,pk表示D中第k类样本所占的比例。根据其定义,Ent的值越小,信息纯度越高。Ent的范围是[0,log|Y|]
下面要选择某个属性进行划分,要依次考虑每个属性,假设当前考虑属性a,a的取值有|V|种,那么我们希望取a作为划分属性,划分到|V|个子节点后,所有子节点的信息熵之和即划分后的信息熵能够有很大的减小,减小的最多的那个属性就是我们选择的属性。
划分后的信息熵定义为:$$/sum_{v=1}^V{|D^v| /over |D|}Ent(D^v) $$
所以用属性a对样本集D进行划分的信息增益就是原来的信息熵减去划分后的信息熵:$$Gain(D,a) = Ent(D) - /sum_{v=1}^V{|D^v| /over |D|}Ent(D^v) $$
ID3算法就是这样每次选择一个属性对样本集进行划分,知道两种情况使这个过程停止:
(1)某个子节点样本全部属于一类
(2)属性都用完了,这时候如果子节点样本还是不一致,那么只好少数服从多数了
ID3算法实现(纯标称值)
如果样本全部是标称值即离散值的话,会比较简单。
代码:
#决策树生成算法: treesID3.py
from math import log
from operator import itemgetter
def createDataSet(): #创建数据集
dataSet = [[1,1,'yes'],
[1,1,'yes'],
[1,0,'no'],
[0,1,'no'],
[0,1,'no']]
featname = ['no surfacing', 'flippers']
return dataSet,featname
def filetoDataSet(filename):
fr = open(filename,'r')
all_lines = fr.readlines()
featname = all_lines[0].strip().split(',')[1:-1]
print(featname)
dataSet = []
for line in all_lines[1:]:
line = line.strip()
lis = line.split(',')[1:]
dataSet.append(lis)
fr.close()
return dataSet,featname
def calcEnt(dataSet): #计算香农熵
numEntries = len(dataSet)
labelCounts = {}
for featVec in dataSet:
label = featVec[-1]
if label not in labelCounts.keys():
labelCounts[label] = 0
labelCounts[label] += 1
Ent = 0.0
for key in labelCounts.keys():
p_i = float(labelCounts[key]/numEntries)
Ent -= p_i * log(p_i,2)
return Ent
def splitDataSet(dataSet, axis, value): #划分数据集,找出第axis个属性为value的数据
returnSet = []
for featVec in dataSet:
if featVec[axis] == value:
retVec = featVec[:axis]
retVec.extend(featVec[axis+1:])
returnSet.append(retVec)
return returnSet
def chooseBestFeat(dataSet):
numFeat = len(dataSet[0])-1
Entropy = calcEnt(dataSet)
DataSetlen = float(len(dataSet))
bestGain = 0.0
bestFeat = -1
for i in range(numFeat):
allvalue = [featVec[i] for featVec in dataSet]
specvalue = set(allvalue)
nowEntropy = 0.0
for v in specvalue:
Dv = splitDataSet(dataSet,i,v)
p = len(Dv)/DataSetlen
nowEntropy += p * calcEnt(Dv)
if Entropy - nowEntropy > bestGain:
bestGain = Entropy - nowEntropy
bestFeat = i
return bestFeat
def Vote(classList):
classdic = {}
for vote in classList:
if vote not in classdic.keys():
classdic[vote] = 0
classdic[vote] += 1
sortedclassDic = sorted(classdic.items(),key=itemgetter(1),reverse=True)
return sortedclassDic[0][0]
def createDecisionTree(dataSet,featnames):
featname = featnames[:] ################
classlist = [featvec[-1] for featvec in dataSet] #此节点的分类情况
if classlist.count(classlist[0]) == len(classlist): #全部属于一类
return classlist[0]
if len(dataSet[0]) == 1: #分完了,没有属性了
return Vote(classlist) #少数服从多数
# 选择一个最优特征进行划分
bestFeat = chooseBestFeat(dataSet)
bestFeatname = featname[bestFeat]
del(featname[bestFeat]) #防止下标不准
DecisionTree = {bestFeatname:{}}
# 创建分支,先找出所有属性值,即分支数
allvalue = [vec[bestFeat] for vec in dataSet]
specvalue = sorted(list(set(allvalue))) #使有一定顺序
for v in specvalue:
copyfeatname = featname[:]
DecisionTree[bestFeatname][v] = createDecisionTree(splitDataSet(dataSet,bestFeat,v),copyfeatname)
return DecisionTree
if __name__ == '__main__':
filename = "D://MLinAction//Data//西瓜2.0.txt"
DataSet,featname = filetoDataSet(filename)
Tree = createDecisionTree(DataSet,featname)
print(Tree)
解释一下几个函数:
filetoDataSet(filename) 将文件中的数据整理成数据集
calcEnt(dataSet) 计算香农熵
splitDataSet(dataSet, axis, value) 划分数据集,选择出第axis个属性的取值为value的所有数据集,即D^v,并去掉第axis个属性,因为不需要了
chooseBestFeat(dataSet) 根据信息增益,选择一个最好的属性
Vote(classList) 如果属性用完,类别仍不一致,投票决定
createDecisionTree(dataSet,featnames) 递归创建决策树
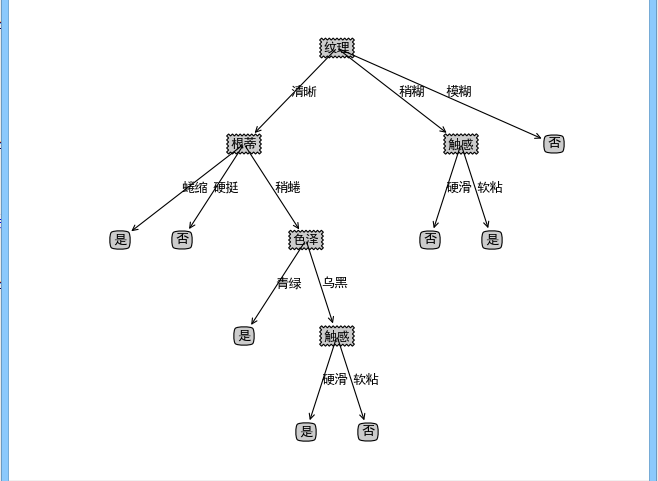
用西瓜数据集2.0对算法进行测试,西瓜数据集见西瓜数据集2.0,输出如下:
['色泽', '根蒂', '敲声', '纹理', '脐部', '触感']
{'纹理': {'清晰': {'根蒂': {'蜷缩': '是', '硬挺': '否', '稍蜷': {'色泽': {'青绿': '是', '乌黑': {'触感': {'硬滑': '是', '软粘': '否'}}}}}}, '稍糊': {'触感': {'硬滑': '否', '软粘': '是'}}, '模糊': '否'}}
 由于数据太少,没有设置测试数据以验证其准确度,但是我后面会根据乳腺癌的例子进行准确度的测试的,下面进入下一部分:
由于数据太少,没有设置测试数据以验证其准确度,但是我后面会根据乳腺癌的例子进行准确度的测试的,下面进入下一部分:
有连续值的情况
有连续值的情况如西瓜数据集3.0
一个属性有很多种取值,我们肯定不能每个取值都做一个分支,这时候需要对连续属性进行离散化,有几种方法供选择,其中两种是:
1.对每一类别的数据集的连续值取平均值,再取各类的平均值的平均值作为划分点,将连续属性化为两类变成离散属性
2.C4.5采用的二分法,排序离散属性,取每两个的中点作为划分点的候选点,计算以每个划分点划分数据集的信息增益,取最大的那个划分点将连续属性化为两类变成离散属性,用该属性进行划分的信息增益就是刚刚计算的最大信息增益。公式如下:
$$Gain(D,a) = /max_{t /in T a}Ent(D) - /sum {/lambda /in {+,-}}{|D_t^/lambda| /over |D|}Ent(D_t^/lambda) $$
这里采用第二种,并在学习前对连续属性进行离散化。增加处理的代码如下:
def splitDataSet_for_dec(dataSet, axis, value, small):
returnSet = []
for featVec in dataSet:
if (small and featVec[axis] <= value) or ((not small) and featVec[axis] > value):
retVec = featVec[:axis]
retVec.extend(featVec[axis+1:])
returnSet.append(retVec)
return returnSet
def DataSetPredo(filename,decreteindex):
dataSet,featname = filetoDataSet(filename)
Entropy = calcEnt(dataSet)
DataSetlen = len(dataSet)
for index in decreteindex: #对每一个是连续值的属性下标
for i in range(DataSetlen):
dataSet[i][index] = float(dataSet[i][index])
allvalue = [vec[index] for vec in dataSet]
sortedallvalue = sorted(allvalue)
T = []
for i in range(len(allvalue)-1): #划分点集合
T.append(float(sortedallvalue[i]+sortedallvalue[i+1])/2.0)
bestGain = 0.0
bestpt = -1.0
for pt in T: #对每个划分点
nowent = 0.0
for small in range(2): #化为正类负类
Dt = splitDataSet_for_dec(dataSet, index, pt, small)
p = len(Dt) / float(DataSetlen)
nowent += p * calcEnt(Dt)
if Entropy - nowent > bestGain:
bestGain = Entropy-nowent
bestpt = pt
featname[index] = str(featname[index]+"<="+"%.3f"%bestpt)
for i in range(DataSetlen):
dataSet[i][index] = "是" if dataSet[i][index] <= bestpt else "否"
return dataSet,featname
输出处理过后的属性以及对西瓜数据集3.0的输出
['色泽', '根蒂', '敲声', '纹理', '脐部', '触感', '密度<=0.382', '含糖率<=0.126']
{'纹理': {'模糊': '否', '稍糊': {'触感': {'硬滑': '否', '软粘': '是'}}, '清晰': {'密度<=0.382': {'是': '否', '否': '是'}}}}
主要是预处理函数DataSetPredo,对数据集提前离散化,然后再进行学习,学习代码类似。输出的决策树如下:

有缺失值的情况
数据有缺失值是常见的情况,我们不好直接抛弃这些数据,因为这样会损失大量数据,不划算,但是缺失值我们也无法判断它的取值。怎么办呢,办法还是有的。
考虑两个问题:
1.有缺失值时如何进行划分选择
2.已选择划分属性,有缺失值的样本划不划分,如何划分?
基本思想是进行最优属性选择时,先只考虑无缺失值样本,然后再乘以相应比例,得到在整个样本集上的大致情况。连带考虑到第二个问题的话,考虑给每一个样本一个权重,此时每个样本不再总是被看成一个独立样本,这样有利于第二个问题的解决:即若样本在属性a上的值缺失,那么将其看成是所有值都取,只不过取每个值的权重不一样,每个值的权重参考该值在无缺失值样本中的比例,简单地说,比如在无缺失值样本集中,属性a取去两个值1和2,并且取1的权重和占整个权重和1/3,而取2的权重和占2/3,那么依据该属性对样本集进行划分时,遇到该属性上有缺失值的样本,那么我们认为该样本取值2的可能性更大,于是将该样本的权重乘以2/3归到取值为2的样本集中继续进行划分构造决策树,而乘1/3划到取值为1的样本集中继续构造。不知道我说清楚没有。
公式如下:

其中,D~表示数据集D在属性a上无缺失值的样本,根据它来判断a属性的优劣,rho(即‘lou’)表示属性a的无缺失值样本占所有样本的比例,p~_k表示无缺失值样本中第k类所占的比例,r~_v表示无缺失值样本在属性a上取值为v的样本所占的比例。
在划分样本时,如果有缺失值,则将样本划分到所有子节点,在属性a取值v的子节点上的权重为r~_v * 原来的权重。
更详细的解读参考《机器学习》P86-87。
根据权重法修改后的ID3算法实现如下:
#修改 treesID3.py
from math import log
from operator import itemgetter
def filetoDataSet(filename):
fr = open(filename,'r')
all_lines = fr.readlines()
featname = all_lines[0].strip().split(',')[1:-1]
dataSet = []
for line in all_lines[1:]:
line = line.strip()
lis = line.split(',')[1:]
dataSet.append(lis)
fr.close()
return dataSet,featname
def calcEnt(dataSet, weight): #计算权重香农熵
labelCounts = {}
i = 0
for featVec in dataSet:
label = featVec[-1]
if label not in labelCounts.keys():
labelCounts[label] = 0
labelCounts[label] += weight[i]
i += 1
Ent = 0.0
for key in labelCounts.keys():
p_i = float(labelCounts[key]/sum(weight))
Ent -= p_i * log(p_i,2)
return Ent
def splitDataSet(dataSet, weight, axis, value, countmissvalue): #划分数据集,找出第axis个属性为value的数据
returnSet = []
returnweight = []
i = 0
for featVec in dataSet:
if featVec[axis] == '?' and (not countmissvalue):
continue
if countmissvalue and featVec[axis] == '?':
retVec = featVec[:axis]
retVec.extend(featVec[axis+1:])
returnSet.append(retVec)
if featVec[axis] == value:
retVec = featVec[:axis]
retVec.extend(featVec[axis+1:])
returnSet.append(retVec)
returnweight.append(weight[i])
i += 1
return returnSet,returnweight
def splitDataSet_for_dec(dataSet, axis, value, small, countmissvalue):
returnSet = []
for featVec in dataSet:
if featVec[axis] == '?' and (not countmissvalue):
continue
if countmissvalue and featVec[axis] == '?':
retVec = featVec[:axis]
retVec.extend(featVec[axis+1:])
returnSet.append(retVec)
if (small and featVec[axis] <= value) or ((not small) and featVec[axis] > value):
retVec = featVec[:axis]
retVec.extend(featVec[axis+1:])
returnSet.append(retVec)
return returnSet
def DataSetPredo(filename,decreteindex): #首先运行,权重不变为1
dataSet,featname = filetoDataSet(filename)
DataSetlen = len(dataSet)
Entropy = calcEnt(dataSet,[1 for i in range(DataSetlen)])
for index in decreteindex: #对每一个是连续值的属性下标
UnmissDatalen = 0
for i in range(DataSetlen): #字符串转浮点数
if dataSet[i][index] != '?':
UnmissDatalen += 1
dataSet[i][index] = float(dataSet[i][index])
allvalue = [vec[index] for vec in dataSet if vec[index] != '?']
sortedallvalue = sorted(allvalue)
T = []
for i in range(len(allvalue)-1): #划分点集合
T.append(float(sortedallvalue[i]+sortedallvalue[i+1])/2.0)
bestGain = 0.0
bestpt = -1.0
for pt in T: #对每个划分点
nowent = 0.0
for small in range(2): #化为正类(1)负类(0)
Dt = splitDataSet_for_dec(dataSet, index, pt, small, False)
p = len(Dt) / float(UnmissDatalen)
nowent += p * calcEnt(Dt)
if Entropy - nowent > bestGain:
bestGain = Entropy-nowent
bestpt = pt
featname[index] = str(featname[index]+"<="+"%.3f"%bestpt)
for i in range(DataSetlen):
if dataSet[i][index] != '?':
dataSet[i][index] = "是" if dataSet[i][index] <= bestpt else "否"
return dataSet,featname
def getUnmissDataSet(dataSet, weight, axis):
returnSet = []
returnweight = []
tag = []
i = 0
for featVec in dataSet:
if featVec[axis] == '?':
tag.append(i)
else:
retVec = featVec[:axis]
retVec.extend(featVec[axis+1:])
returnSet.append(retVec)
i += 1
for i in range(len(weight)):
if i not in tag:
returnweight.append(weight[i])
return returnSet,returnweight
def printlis(lis):
for li in lis:
print(li)
def chooseBestFeat(dataSet,weight,featname):
numFeat = len(dataSet[0])-1
DataSetWeight = sum(weight)
bestGain = 0.0
bestFeat = -1
for i in range(numFeat):
UnmissDataSet,Unmissweight = getUnmissDataSet(dataSet, weight, i) #无缺失值数据集及其权重
Entropy = calcEnt(UnmissDataSet,Unmissweight) #Ent(D~)
allvalue = [featVec[i] for featVec in dataSet if featVec[i] != '?']
UnmissSumWeight = sum(Unmissweight)
lou = UnmissSumWeight / DataSetWeight #lou
specvalue = set(allvalue)
nowEntropy = 0.0
for v in specvalue: #该属性的几种取值
Dv,weightVec_v = splitDataSet(dataSet,Unmissweight,i,v,False) #返回 此属性为v的所有样本 以及 每个样本的权重
p = sum(weightVec_v) / UnmissSumWeight #r~_v = D~_v / D~
nowEntropy += p * calcEnt(Dv,weightVec_v)
if lou*(Entropy - nowEntropy) > bestGain:
bestGain = Entropy - nowEntropy
bestFeat = i
return bestFeat
def Vote(classList,weight):
classdic = {}
i = 0
for vote in classList:
if vote not in classdic.keys():
classdic[vote] = 0
classdic[vote] += weight[i]
i += 1
sortedclassDic = sorted(classdic.items(),key=itemgetter(1),reverse=True)
return sortedclassDic[0][0]
def splitDataSet_adjustWeight(dataSet,weight,axis,value,r_v):
returnSet = []
returnweight = []
i = 0
for featVec in dataSet:
if featVec[axis] == '?':
retVec = featVec[:axis]
retVec.extend(featVec[axis+1:])
returnSet.append(retVec)
returnweight.append(weight[i] * r_v)
elif featVec[axis] == value:
retVec = featVec[:axis]
retVec.extend(featVec[axis+1:])
returnSet.append(retVec)
returnweight.append(weight[i])
i += 1
return returnSet,returnweight
def createDecisionTree(dataSet,weight,featnames):
featname = featnames[:] ################
classlist = [featvec[-1] for featvec in dataSet] #此节点的分类情况
if classlist.count(classlist[0]) == len(classlist): #全部属于一类
return classlist[0]
if len(dataSet[0]) == 1: #分完了,没有属性了
return Vote(classlist,weight) #少数服从多数
# 选择一个最优特征进行划分
bestFeat = chooseBestFeat(dataSet,weight,featname)
bestFeatname = featname[bestFeat]
del(featname[bestFeat]) #防止下标不准
DecisionTree = {bestFeatname:{}}
# 创建分支,先找出所有属性值,即分支数
allvalue = [vec[bestFeat] for vec in dataSet if vec[bestFeat] != '?']
specvalue = sorted(list(set(allvalue))) #使有一定顺序
UnmissDataSet,Unmissweight = getUnmissDataSet(dataSet, weight, bestFeat) #无缺失值数据集及其权重
UnmissSumWeight = sum(Unmissweight) # D~
for v in specvalue:
copyfeatname = featname[:]
Dv,weightVec_v = splitDataSet(dataSet,Unmissweight,bestFeat,v,False) #返回 此属性为v的所有样本 以及 每个样本的权重
r_v = sum(weightVec_v) / UnmissSumWeight #r~_v = D~_v / D~
sondataSet,sonweight = splitDataSet_adjustWeight(dataSet,weight,bestFeat,v,r_v)
DecisionTree[bestFeatname][v] = createDecisionTree(sondataSet,sonweight,copyfeatname)
return DecisionTree
if __name__ == '__main__':
filename = "D://MLinAction//Data//西瓜2.0alpha.txt"
DataSet,featname = DataSetPredo(filename,[])
Tree = createDecisionTree(DataSet,[1.0 for i in range(len(DataSet))],featname)
print(Tree)
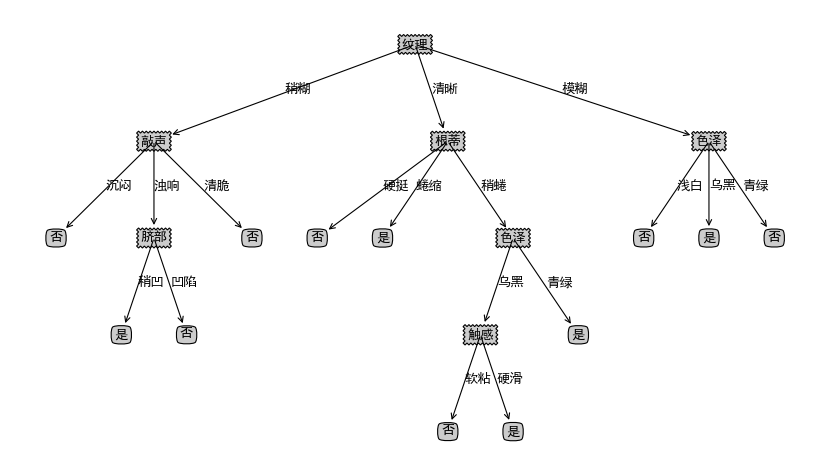
有缺失值的情况如西瓜数据集2.0alpha
实验结果:
{'纹理': {'模糊': {'色泽': {'青绿': '否', '浅白': '否', '乌黑': '是'}}, '清晰': {'根蒂': {'蜷缩': '是', '硬挺': '否', '稍蜷': {'色泽': {'青绿': '是', '乌黑': {'触感': {'硬滑': '是', '软粘': '否'}}}}}}, '稍糊': {'敲声': {'浊响': {'脐部': {'凹陷': '否', '稍凹': '是'}}, '清脆': '否', '沉闷': '否'}}}}
在乳腺癌数据集上的测试与表现
有了算法,我们当然想做一定的测试看一看算法的表现。这里我选择了威斯康辛女性乳腺癌的数据。
数据总共有9列,每一列分别代表,以逗号分割
1 Sample code number (病人ID)
2 Clump Thickness 肿块厚度
3 Uniformity of Cell Size 细胞大小的均匀性
4 Uniformity of Cell Shape 细胞形状的均匀性
5 Marginal Adhesion 边缘粘
6 Single Epithelial Cell Size 单上皮细胞的大小
7 Bare Nuclei 裸核
8 Bland Chromatin 乏味染色体
9 Normal Nucleoli 正常核
10 Mitoses 有丝分裂
11 Class: 2 for benign, 4 formalignant(恶性或良性分类)
[from Toby]
总共700条左右的数据,选取最后80条作为测试集,前面作为训练集,进行学习。
使用分类器的代码如下:
import treesID3 as id3
import treePlot as tpl
import pickle
def classify(Tree, featnames, X):
classLabel = "未知"
root = list(Tree.keys())[0]
firstGen = Tree[root]
featindex = featnames.index(root) #根节点的属性下标
for key in firstGen.keys(): #根属性的取值,取哪个就走往哪颗子树
if X[featindex] == key:
if type(firstGen[key]) == type({}):
classLabel = classify(firstGen[key],featnames,X)
else:
classLabel = firstGen[key]
return classLabel
def StoreTree(Tree,filename):
fw = open(filename,'wb')
pickle.dump(Tree,fw)
fw.close()
def ReadTree(filename):
fr = open(filename,'rb')
return pickle.load(fr)
if __name__ == '__main__':
filename = "D://MLinAction//Data//breastcancer.txt"
dataSet,featnames = id3.DataSetPredo(filename,[0,1,2,3,4,5,6,7,8])
Tree = id3.createDecisionTree(dataSet[:620],[1.0 for i in range(len(dataSet))],featnames)
tpl.createPlot(Tree)
storetree = "D://MLinAction//Data//decTree.dect"
StoreTree(Tree,storetree)
#Tree = ReadTree(storetree)
i = 1
cnt = 0
for lis in dataSet[620:]:
judge = classify(Tree,featnames,lis[:-1])
shouldbe = lis[-1]
if judge == shouldbe:
cnt += 1
print("Test %d was classified %s, it's class is %s %s" %(i,judge,shouldbe,"=====" if judge==shouldbe else ""))
i += 1
print("The Tree's Accuracy is %.3f" % (cnt / float(i)))
结果(点击右键看大图)
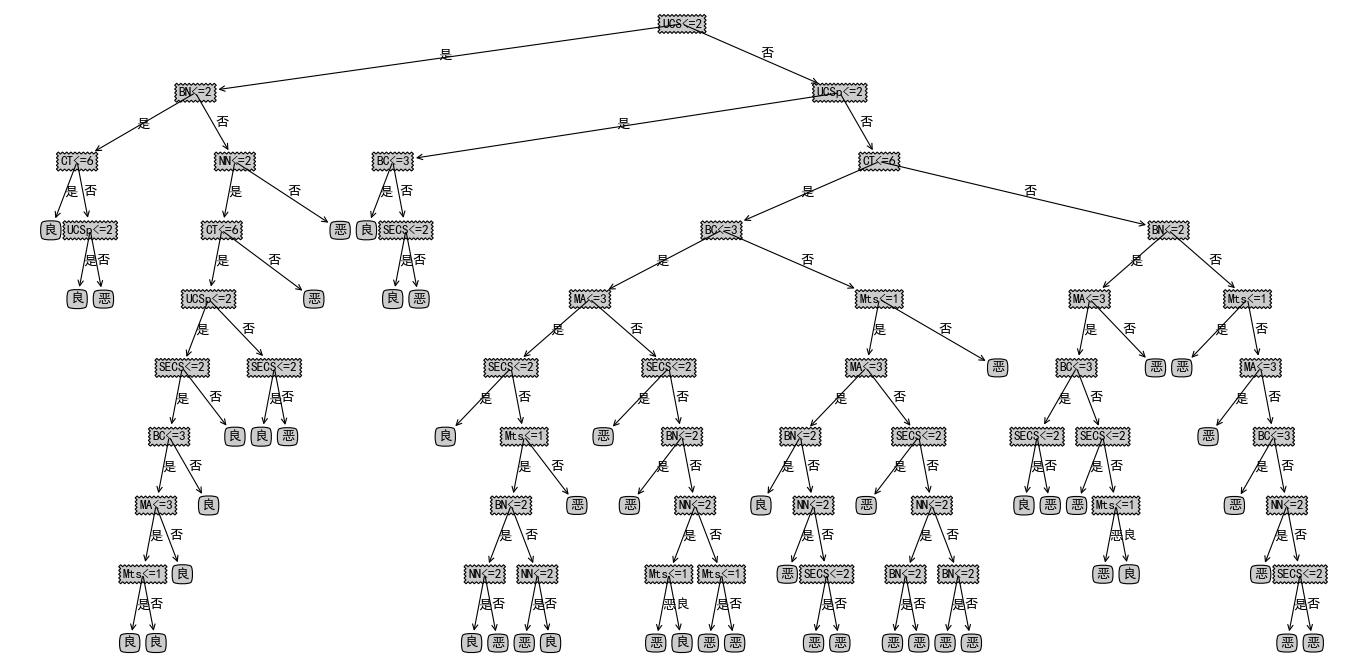
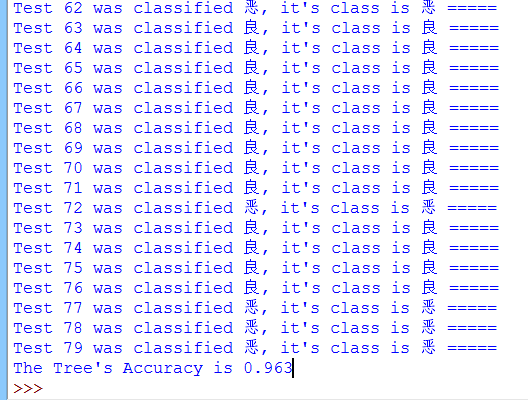
正确率约为96%左右,算是不差的分类器了。
我的乳腺癌数据见: 乳腺癌数据
至此,决策树算法ID3的实现完毕,下面考虑基于基尼指数和信息增益率进行划分选择,以及考虑实现剪枝过程,因为我们可以看到上面训练出的决策树还存在着很多冗余分支,是因为实现过程中,由于数据量太大,每个分支都不完全纯净,所以会创建往下的分支,但是分支投票的结果又是一致的,而且数据量再大,特征数再多的话,决策树会非常大非常复杂,所以剪枝一般是必做的一步。剪枝分为先剪枝和后剪枝,如果细说的话可以写很多了。
利用matplotlib画决策树的代码见 利用matplotlib画决策树
此文亦可见: Here
MathJax的一些使用方法见 1 2 3
参考资料:《机器学习》《机器学习实战》通过本次实战也发现了这两本书中的一些错误之处。
lz初学机器学习不久,如有错漏之处请多包涵指出或者各位有什么想法或意见欢迎评论去告诉我:-)









![[HBLOG]公众号](http://www.liuhaihua.cn/img/qrcode_gzh.jpg)

