谷歌《Cell》论文:使用深度学习,直接对细胞影像生成荧光标记
开源地址:https://github.com/google/in-silico-labeling
在生物学和医学领域,显微镜技术可以帮助研究人员观察到肉眼看不到的细胞和分子的细节。透射光学显微镜将生物样本的一侧照射并成像,这种显微镜相对简单,活体培养物可以很好地耐受,但是生成的图像可能难以准确评估。荧光显微镜可以将需要的生物对象(如细胞核)用荧光分子做出特殊标记,简化分析,但需要复杂的样品制备。随着机器学习在显微镜学中的应用越发广泛(包括用于自动评估图像质量和帮助病理学家诊断癌组织的算法),谷歌的研究者开始思考,是否可以开发一个深度学习系统,将两种显微镜技术的优点结合起来,同时最大限度地克服二者的缺点?
今天,谷歌在《细胞》(Cell)杂志上发表了一篇名为《In Silico Labeling: Predicting Fluorescent Labels in Unlabeled Images》的文章。在论文中,研究者指出,深层神经网络可以从透射光图像预测荧光图像,在不对细胞做出改变的情况下生成标记的有用图像,同时有可能实现未修饰细胞的纵向研究、用于细胞治疗的微创细胞筛选以及使用大量同时标记的研究。此外,谷歌还开源了模型、完整的训练和测试数据、经过培训的模型检查点及示例代码。
背景
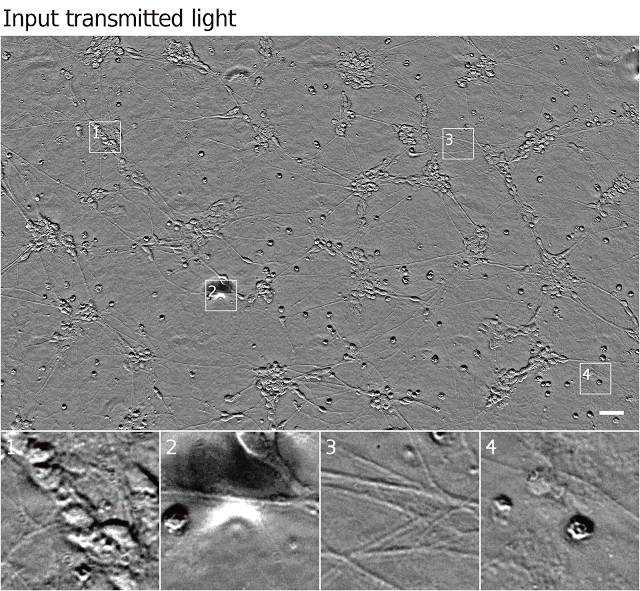
透射光显微镜技术用法简单,但根据该显微镜生成的图像很难判断细胞的情况。下图中的示例图像来自相差显微镜,图中像素的强度表明光在通过样本时相移的程度。
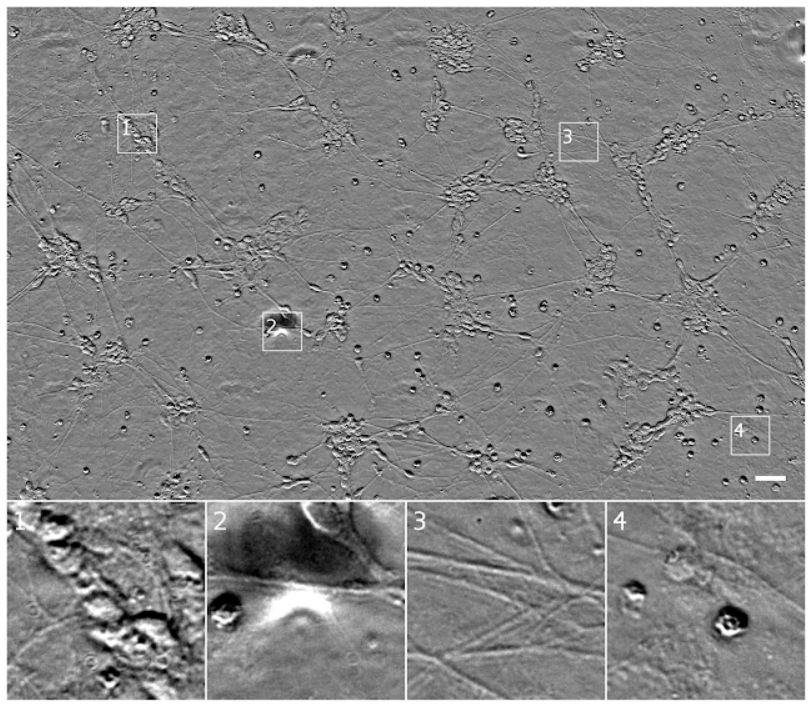
来自诱导性多能干细胞的人类运动神经元培养物的透射光(相差显微)图像。Outset 1 展示了一群细胞,可能是神经元。Outset 2 展示了图像中的一个裂痕,它遮挡了底层的细胞。Outset 3 展示了神经轴突。Outset 4 展示了细胞死亡。上图比例尺为 40 μm。本文图源:格莱斯顿研究所(Gladstone Institutes)史蒂夫·芬克拜纳实验室(Finkbeiner lab)。
在上图中,我们很难确定 Outset 1 的集群中的细胞数量或 Outset 4(提示:中上部分有一个几乎不可见的扁平细胞)中的细胞状态与位置,同样也很难获得始终清晰的精细结构,如 Outset 3 中的神经突触。
我们可以通过在 z 堆栈的维度上收集图像,从透射光显微镜中获取更多信息,其中 z 堆栈表示相机距离且会系统地变化,而(x, y)表示注册的图像集。这会令细胞的不同部分分别进入或脱离焦点,且提供了样本的 3D 结构信息。不幸的是,这种方法通常需要已训练的系统来理解 z 堆栈,并且 z 堆栈的分析目前已基本实现自动化。z 堆栈的案例如下所示。
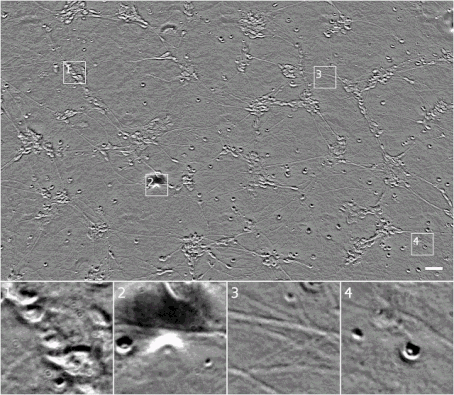
相同细胞 z 堆栈的相差显微镜图。请注意在焦点转移时外观是如何变化的。现在,我们可以看到,Outset 1 右下角的模糊形状是一个椭圆细胞,Outset 4 最右边的细胞要比最上面的细胞高,这可能意味着它已经历了细胞程序化凋亡。
相比之下,荧光显微镜图像更容易分析,因为样本是用荧光标记制备的,这些标记展示了研究者希望查看的信息。例如大多数人的细胞只有一个细胞核,因此标记后的细胞核(下图蓝色部分)可用简单的工具找到并在图像中计数细胞数量。
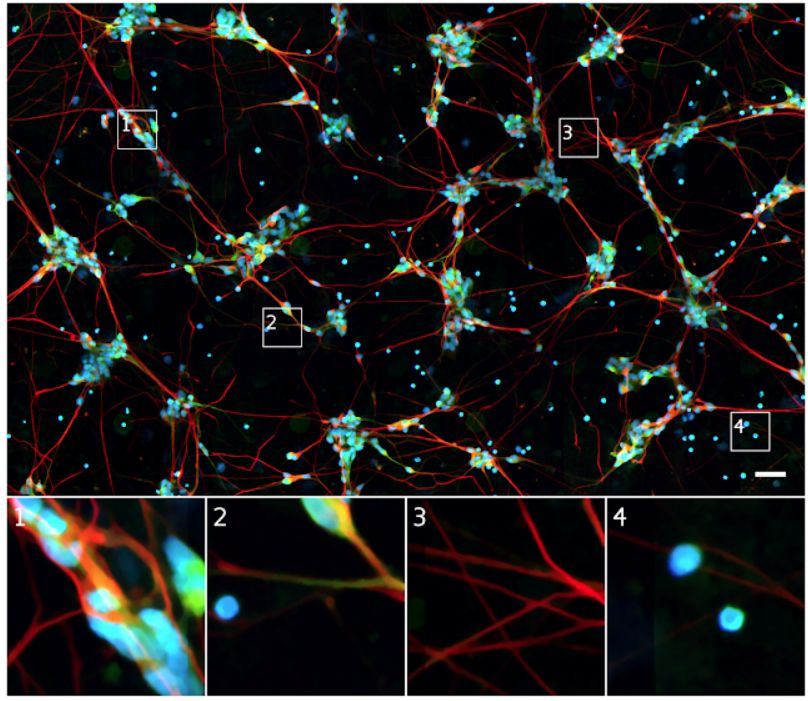
同一细胞的荧光显微镜图像。蓝色荧光集中标记了 DNA,展示了细胞核的位置。绿色荧光集中标记了仅在树突(一种神经细胞子结构)中存在的蛋白质。红色荧光集中标记了仅在轴突(另一种神经细胞子结构)中存在的蛋白质。使用这些标记可以更简单地理解样本中发生的变化。例如,Outset 1 中绿色和红色标记确定其为一个神经集群。Outset 3 中的红色标记表示神经突触是轴突而不是树突。Outset 4 左上角的蓝色点表示这里存在以前难以看到的细胞核,而左侧缺乏蓝色标记的细胞表示它是没有 DNA 的细胞碎片。
然而,荧光显微镜可能存在显著的缺点。首先,样本制备和荧光标记会带来新的复杂性和不可控变量。其次,如果样本中存在很多不同的荧光标记,光谱的重叠会令我们很难判断哪种颜色属于哪个标记,这也就限制了研究人员在一个样本中通常只使用三到四种荧光标记。最后,对于细胞来说,荧光标记可能具有毒性,有时甚至会直接杀死细胞。这使得标记的使用在纵向研究中存在困难,因为纵向研究需要对同一群细胞进行跟踪观察。
利用深度学习看到更多
我们在论文中展示了深度神经网络可以利用透射光 z-堆栈预测荧光图像。为此,我们创建了一个与荧光图像匹配的透射光 z-堆栈数据集,并训练神经网络利用透射光 z-堆栈预测荧光图像。下图展示了该过程:
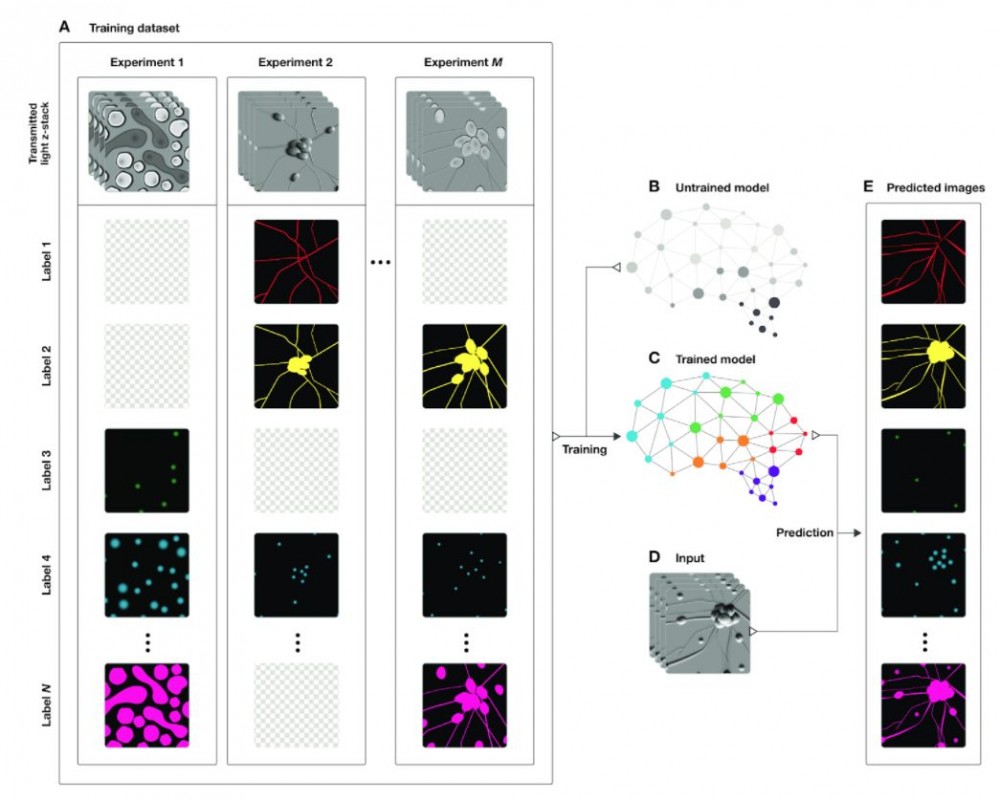
系统概览。(A)训练样本数据集:成对透射光 z-stack 和相同场景的荧光图像的像素配准集合。多个不同的荧光标签用于生成荧光图像,不同训练样本的荧光标签不同;棋盘格图像表示给定样本未获取的荧光标签。(B)未训练深度网络。(C)该网络在数据 A 上训练。(D)新场景图像的 z-堆栈。(E)训练好的网络 C,用于预测新图像 D 中每个像素的荧光标签(标签从数据 A 中学得)。
研究过程中,谷歌开发了一种新型神经网络,该网络受 Inception 的模块设计启发,包括三种基本构造块:in-scale 配置,不改变特征的空间尺度;down-scale 配置,将空间尺度放大一倍;up-scale 配置,将空间尺度缩小 1/2。这帮助我们将网络架构设计这个难题分阶层两个简单一些的问题:构造块的排列(宏观),以及构造块本身的设计(微观)。研究者使用论文讨论的设计原则解决了第一个问题,使用 Google Hypertune 支持的自动搜索功能解决了第二个问题。
为了确保该方法有效,研究者使用 Alphabet 实验室和两个外部伙伴(格莱斯顿研究所史蒂夫·芬克拜纳实验室和哈佛 Rubin 实验室)的数据对模型进行了验证。这些数据跨越三种透射光图像模式(明视野、相位对比和微分干涉相位差)和三种培养类型(诱导性多能干细胞形成的人类运动神经元、大鼠皮层培养和人类乳腺癌细胞)。他们发现该方法能够准确预测多种标签,包括细胞核、细胞类型(如神经细胞)和细胞状态(如细胞死亡)。下图展示了该模型对透射光输入的预测结果以及运动神经元样本的真值荧光反应。
上画展示了相同细胞的透射光图像、荧光图像,以及用谷歌的模型预测的荧光标记。Outset 2 表明尽管输入图像中有伪影,该模型也可以预测正确的标记。Outset 3 表明模型推断出这些结构是轴突,可能是因为它们离最近邻的细胞比较远。Outset 4 表明模型可以发现顶部很难察觉的细胞,并正确地将左侧的目标识别为无 DNA 的细胞碎片。
自己动手尝试一下吧!
谷歌已经开源了模型、完整的数据集、训练和推断代码,以及示例。只需要用最小量的额外训练数据,模型就可以预测新的标记:在论文和代码中,谷歌甚至展示了只需要单张图像就可以预测新标记的结果,这是迁移学习的威力。
谷歌希望,这种不需要干预细胞就能生成有用的标记图像的应用可以在生物学和医学中打开全新的实验领域。
论文:In Silico Labeling: Predicting Fluorescent Labels in Unlabeled Images

论文地址:http://www.cell.com/cell/fulltext/S0092-8674(18)30364-7
摘要:显微术在生命科学中是核心方法。很多常用的方法例如抗体标记等被用于给细胞成分加上物理荧光标记。然而,这些方法有明显的缺点,包括不一致性、由于空间重叠导致能同时标记的数量有限,以及为生成测量数据实验中必然存在的干扰(如细胞固定等)。我们在本文中证明,机器学习方法「in silico labeling(ISL)」能可靠地从未标记的固定样本或活体样本的透射光图像中预测某些荧光标记。ISL 可以预测多种标记,包括对细胞核、细胞类型(如神经细胞)和细胞状态(如细胞死亡)的标记。由于预测是基于计算机的,因此该方法是一致的,不会受限于空间重叠,并且不会对实验造成干扰。ISL 可以生成用其它方法很难或无法观测的生物学测量数据。
原文链接: https://research.googleblog.com/2018/04/seeing-more-with-in-silico-labeling-of_12.html










![[HBLOG]公众号](http://www.liuhaihua.cn/img/qrcode_gzh.jpg)

