背景
做实验的时候,需要将独立测试集的数据与别人server跑出来的结果进行比较,比如下面这个: http://bioinfo.ggc.org/bindn/ 。但是这个server一次性只能提交一个fasta文件,也就是说,我有很多数据的话,就要分多次提交。如果是人工的去操作,会比较耗时,而且工作量特别大,因此这里就需要模拟网页的数据提交。这就是本文的主要内容,
思路
下面先来理清下思路。我的目的是通过自己构造post数据来实现数据提交。
当模拟在网页上提交数据时,首先要弄清楚整个数据处理流程,比如发送了什么样的数据,给谁发的等。那么如果我要在网页上提交数据的话,肯定是要传递参数的,所以我们要知道如何查找这些参数,这是最重要的一点。其次,模拟数据提交,必须要知道提交前的网页和提交后的网页,这样才能将提交后显示结果网页保存下来。最后就是数据处理了,使用正则表达式将需要的数据抽取出来。
实践
参数分析
关于参数,可以从数据包中分析出来,我是使用google自带的抓包工具分析的,使用ctrl+shift+I快捷键,点击进入Network列,如下图:
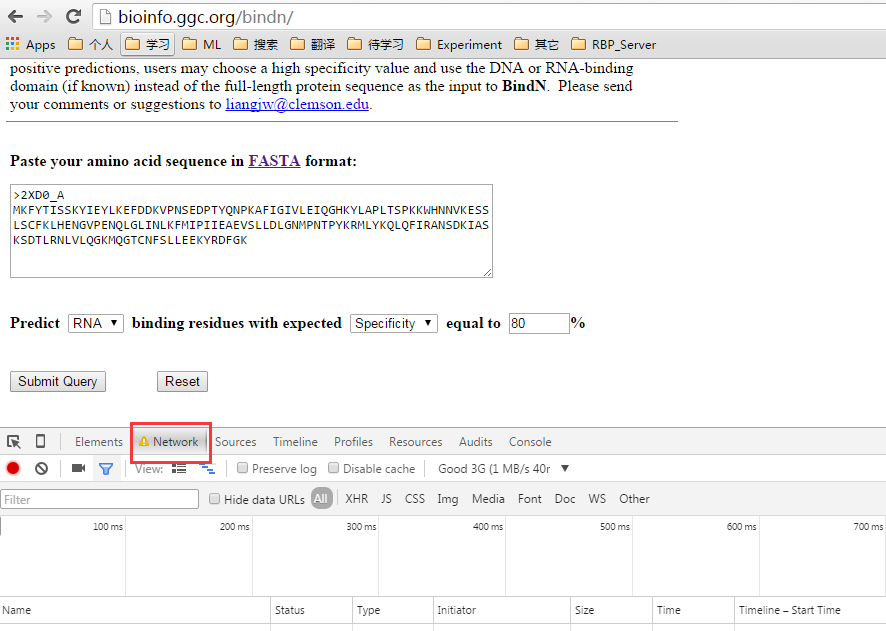
可以看到,当前什么都没有,下面我将参数填写完整

当我将数据设置好之后,点击Submit Query按钮后,结果如下图所示:

多了一个bindn.pl文件,我们来看看这个文件的内容,看看headers部分:

和图二进行比较,你会看到是相互对应。也就是说,这就是我们需要提交的参数:
postData = {'seq' : oneseq, #oneseq是一个字符串,后面作为一个参数传递进来
'qtype' : 'rna',
'vtype' : 'sp',
'val' : '80',
'submit' : 'Submit Query'
}
而点击发送后的请求URL和HTML头内容,如下图:

所以现在我们可以得到以下这些数据(postData在上面已经分析出来了):
hosturl = 'http://bioinfo.ggc.org/bindn/'
posturl = 'http://bioinfo.ggc.org/cgi-bin/bindn/bindn.pl' #可以从数据包中分析出,处理post请求的url
headers = {'User-Agent' : 'Mozilla/5.0 (Windows NT 6.1; WOW64) AppleWebKit/537.36 (KHTML, like Gecko) Chrome/46.0.2490.80 Safari/537.36',
'Referer' : 'http://bioinfo.ggc.org/bindn/'}
Python模拟
分析结束后,我们要构造自己的HTTP数据包,并发送给指定url。我们通过urllib2等几个模块提供的API来实现request请求的发送和相应的接收。最后需要编写一个函数,将自己需要的内容抽取出来。完整代码和讲解如下如下:
# -*- coding: utf-8 -*-
"""
Created on Fri Jan 01 09:34:50 2016
@author: liudiwei
"""
import os
import urllib
import urllib2
import cookielib
import re
#首先定义一个模拟数据提交的函数,传入刚刚分析出来的四个参数即可
def scratchData(hosturl, posturl, postData, headers):
#设置一个cookie处理器,它负责从服务器下载cookie到本地,并且在发送请求时带上本地的cookie
cj = cookielib.LWPCookieJar()
cookie_support = urllib2.HTTPCookieProcessor(cj)
opener = urllib2.build_opener(cookie_support, urllib2.HTTPHandler)
urllib2.install_opener(opener)
#打开登录主页面(他的目的是从页面下载cookie,这样我们在再送post数据时就有cookie了,否则发送不成功)
urllib2.urlopen(hosturl)
#需要给Post数据编码
postDataEncode = urllib.urlencode(postData)
#通过urllib2提供的request方法来向指定Url发送我们构造的数据,并完成数据发送过程
request = urllib2.Request(posturl, postDataEncode, headers)
print request
response = urllib2.urlopen(request)
resultText = response.read()
return resultText
#将一次提交写到一个函数里面,每次只需传入一个序列即可,因为其它的参数不变
def BindN(oneseq, outdir):
#当前页面,即提交数据页面
hosturl = 'http://bioinfo.ggc.org/bindn/'
#post数据接收和处理的页面(我们要向这个页面发送我们构造的Post数据)
posturl = 'http://bioinfo.ggc.org/cgi-bin/bindn/bindn.pl' #可以从数据包中分析出,处理post请求的url
#构造header,一般header至少要包含一下两项。这两项是从抓到的包里分析得出的。
headers = {'User-Agent' : 'Mozilla/5.0 (Windows NT 6.1; WOW64) AppleWebKit/537.36 (KHTML, like Gecko) Chrome/46.0.2490.80 Safari/537.36',
'Referer' : 'http://bioinfo.ggc.org/bindn/'}
#构造Post数据,他也是从抓大的包里分析得出的。
postData = {'seq' : oneseq,
'qtype' : 'rna',
'vtype' : 'sp',
'val' : '80',
'submit' : 'Submit Query'
}
result = scratchData(hosturl, posturl, postData, headers)
print "+++++", oneseq
chainname = oneseq[1:5] + oneseq[6:7]
outfilename = str(chainname) + '.html'
fw_result = open(outdir + '/' + outfilename, 'w')
fw_result.write(result)
fw_result.close()
return result, str(chainname)
#使用正则表达式提取数据
def extractBindN(htmlfmt, outfile):
fw_result = open(outfile, 'w')
inputdata = htmlfmt.split('/n')
for i in range(len(inputdata)):
onedata = inputdata[i].strip()
if not onedata:
continue
if '<' in onedata or '*' in onedata:
continue
regText = onedata.split('/t')[0].strip()
if re.match(r'^/d+$', regText) and True or False:
fw_result.write(onedata + '/n')
fw_result.close()
#main方法
if __name__=="__main__":
oneseq = ">2XD0_A/nMKFYTISSKYIEYLKEFDDKVPNSEDPTYQNPKAFIGIVLEIQGHKYLAPLTSPK/
KWHNNVKESSLSCFKLHENGVPENQLGLINLKFMIPIIEAEVSLLDLGNMPNTPYKRMLYKQLQFIRANSDKIA/
SKSDTLRNLVLQGKMQGTCNFSLLEEKYRDFGK"
outdir = "/home/liudiwei/result" #输出路径
if not os.path.exists(outdir):
os.mkdir(outdir)
print outdir
result, chainname = BindN(oneseq, outdir)
outfile = outdir + "/" + chainname + ".data" #最终输出的文件名
extractBindN(result, outfile)










![[HBLOG]公众号](https://www.liuhaihua.cn/img/qrcode_gzh.jpg)

